微生物单细胞基因组学(SCG)为了解稀有和难培养微生物的基因组提供了手段,是与宏基因组学互补的方法。由于单个微生物细胞中DNA的水平低,因此在进行基因组测序之前需要进行全基因组扩增(WGA)。然而,最常见的WGA方法——多重位点扩增(MDA),十分昂贵且对特定基因组区域有偏差,阻碍了高通量应用,导致基因组覆盖不均匀。因此,从许多物种(尤其是微生物群落中的少数成员)中获得高质量的基因组变得困难。在这里,我们提出了一种体积缩减方法,在标准384孔板中显著降低成本的同时提高了DNA扩增产物的基因组覆盖度和均匀性。我们的结果表明,为获得更高质量的微生物基因组,在专业化和复杂的设置(例如微流体芯片)中进行进一步的体积缩减可能不必要。这种体积缩小方法使得SCG在未来的研究中更加可行,从而有助于我们更广泛地了解环境中那些未被深入研究和未被充分描述的微生物的多样性和功能。
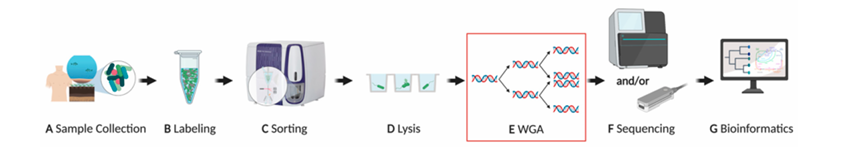
图1 单细胞基因组学的工作流程。
A:获得环境样本后应该立即加工,或在冷冻保护剂的存在下深度冷冻保证细胞的完整性。
B:细胞通常用非特异性荧光染料染色,如 DAPI或SYBR®Green,但它们也可以被特异性标记,例如,荧光原位标记杂化。
C:荧光活化细胞分选(FACS)是最常见的物理分选方法,可将单个细胞分离到多孔板中。
D:一旦单个细胞被分离出来,就会用碱性缓冲液和反复冻融的方法将其破碎,以释放出细胞中的DNA。
E:由于典型的原核细胞DNA含量低,因此需要进行全基因组扩增(WGA)来产生足够的DNA用于文库准备。
F:一旦准备好了DNA文库,就可以分别使用Illumina®和Oxford Nanopore Technologies®等短读/长读测序平台。
G:利用生物信息学进行序列质量评估、组装、分类、ORF呼叫和注释。
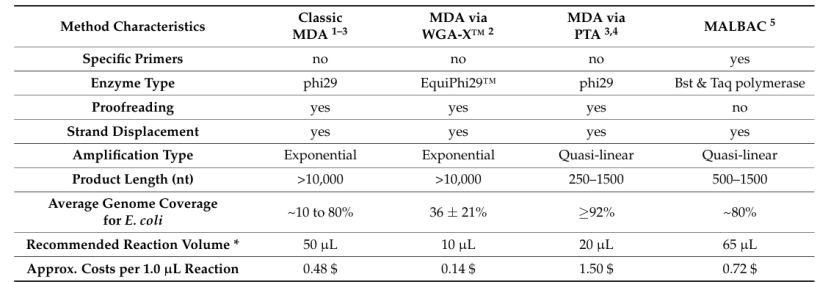
一些微生物体内只含有几飞克DNA,所以其中WGA这一步操作至关重要,为接下来文库的制备和测序奠定基础。近几年来不同的WGA方式也在不断发展,但基于PCR的扩增方法不适用于DNA含量极低的微生物,因此使用phi29高保真酶的MDA方法应运而生,但MDA整体反应的耗费极高,因此使用受限。在这里,我们列举了几种不同的WGA方法。见下表。
表1 微生物单细胞基因组扩增方法的特点
MDA:多重置换扩增;WGA-X:全基因组扩增-X;PTA:原始模板导向扩增;MALBAC:多重退火和循环扩增。
常规的WGA方法反应体系较大,每个反应的花费也较高。因此我们尝试缩小体系。
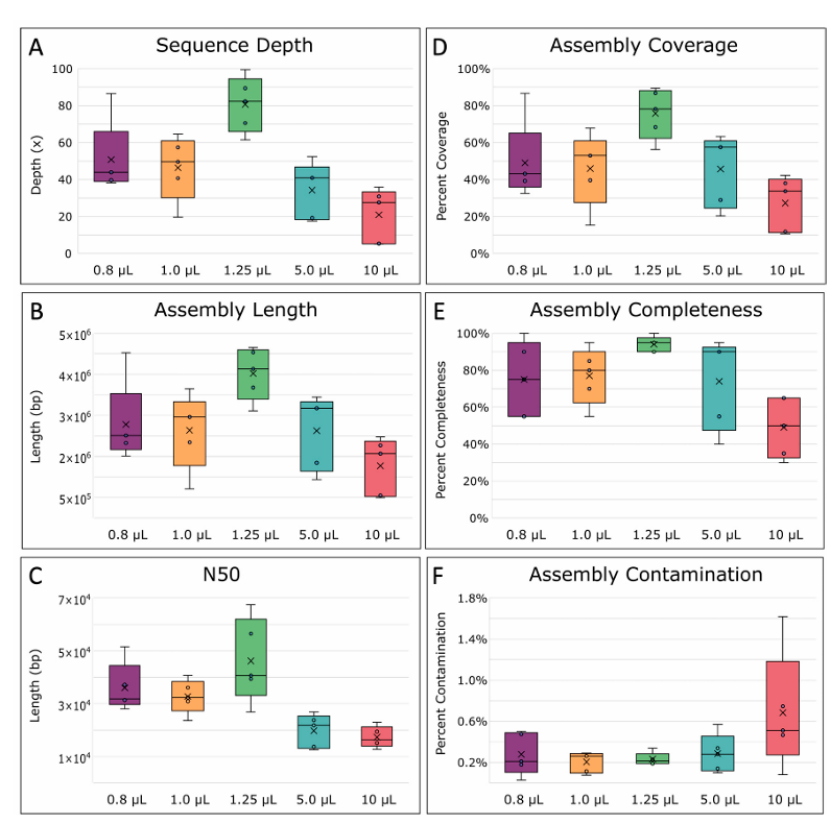
图2 单倍体基因组(SAG)组装统计数据。
(A)最终序列深度,即估计的每个基因组碱基平均测序次数。
(B)组装的总平均长度。
(C)N50平均值,即支持基因组组装50%的最小片段长度。
(D)组装覆盖整个E.oil的百分比。
(E)通过MDMCleaner确定组装基因组的完整性,
(F)组装中的污染碱基百分比,方框中间的线表示中位数,x表示平均值。五重复数据计算。
总体而言,这些结果表明,在1.25 uL的反应体积下进行的MDA操作极大地改进了该方法,产生的SAGs相比于标准的大反应体积,具有更少的偏差、更少的污染和更完整的特性。
采用标准384孔板搭配商用细胞分选机和液体分配器,对其他研究人员来说也很方便。标准50 LMDA反应(表1)这种方法降低了97.5%的成本。研究者初步工作发现WGA-X工作1.25 uL反应量也是如此。本文所做的改进将对其他单细胞研究产生极大的影响,从而增加SCG的使用,特别是用于阐明环境样本中稀有类群和/或新型微生物暗物质的基因组潜力的研究。
在整个工作流程中,分离出单细胞并进行裂解这一步采用了I.DOT非接触式纳升级移液系统。

Dispendix I.DOT非接触式分液系统作为一款纳升级(nl)的微量液体分配设备,采用即时喷点移液技术,系统共有8个独立控制的正压通道。进行液体分配时,在底部具有微米级小孔的源板(Source Plate)上方施加一个特定的压力脉冲,使小孔底部生成一个最小2.55 nl,最大50 nl的液滴,可以释放到目标板(Target Plate)的任意孔中,每个通道每秒最多可生成100个液滴,通过液滴数量的调节,实现从纳升到微升级别的液体分配。
同腾睿杰(上海)生物技有限公司作为CYTENA I.DOT中国总代理商,为您提供优质的售前售后服务。
联系电话:021-50826962
联系邮箱:sales@ttbiotech.com




