在生物生产领域,单克隆性是至关重要的,它要求细胞来自单个细胞且具有遗传同质性。传统的单细胞克隆方法,如有限稀释法,是基于泊松统计概率来估计单克隆性的;这种方法存在诸多弊端,它不仅耗时、通量低,而且需要细胞扩增来确定理想的候选细胞。此外,由于有限稀释可能导致每个孔中有多个细胞,所以往往需要多轮克隆来提高单克隆性的概率[1-3]。

NanoCellect Biomedical, Inc.推出的WOLF G2流式细胞分选仪,它们能够筛选数百万个细胞,并根据多达11个参数对感兴趣的细胞进行特异性分选和单细胞分配,从而满足细胞系开发的需求。本文使用了一种常见的荧光参数—活力染料,来提高细胞的生长。DRAQ7就是一种用于检测细胞活力的远红色生物标志物,它可以与细胞膜受损细胞中的双链DNA(dsDNA)结合 [4,5]。利用配置有488nm和637nm双激光的WOLF G2来分选带有DRAQ7的GFP + HEK293细胞,可以完全排除死细胞,在进行单细胞克隆时最大化铺板密度和细胞的生长[6]。
鞘液和样品缓冲液由1xPBS、0.5% BSA、5mM EDTA和12.5mM HEPES组成。实验设置了多个对照组,包括未染色的HEK293T阴性对照、用DRAQ7染色的活:死(50:50)HEK293T对照以及GFP + HEK293对照。用于分选的样品中含有75%的HEK293T和25%的GFP + HEK293T细胞,并用DRAQ7染色,最终浓度为1.0x10⁵cells/mL。
在分析过程中,应用补偿来创建一个矩阵(图1)。将带有细胞门的散点图应用于FSC-H与FSC-width图以创建单线门。单线门应用于双变量图(DRAQ7 vs FITC)。
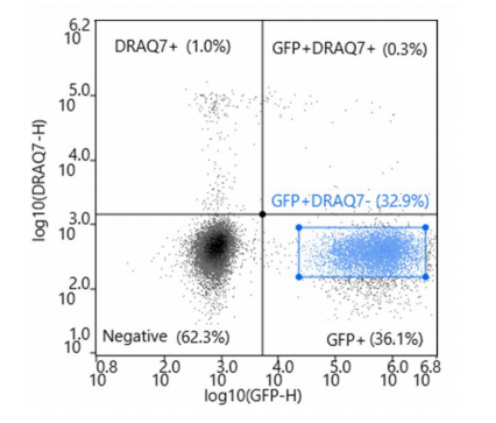
图1. 基于DRAQ7和GFP标记的细胞分选结果分布图
将细胞分选到预先填充有200 uL完整克隆培养基的三个96孔板中,同时准备一个采用有限稀释法(1.0细胞/孔)的96孔板作为对照。这些板在37°C、5% CO₂的环境中培养,并在第0天使用酶标仪的绿色荧光通道检测可能生长成菌落的GFP+细胞。在第7天和第14天,使用明场和绿色荧光通道确认菌落形成并确定细胞生长情况。而且,这个实验重复了三次,每次都使用不同的无菌WOLF分选盒和流体系统。
1、在总体生长情况上,使用配置637nm激光的WOLF G2对带有DRAQ7的GFP+ HEK293细胞进行单细胞分选以排除死细胞时,观察到平均总生长率为81±3.9%,相比有限稀释法的20±8.1%,提高了约4倍(图2)。
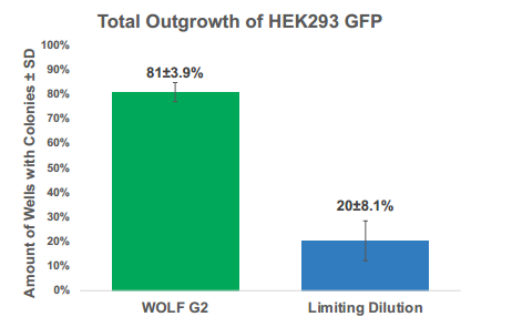
图2. 14天后HEK293 GFP的平均总生长量:与有限稀释法相比,使用WOLF G2用于单细胞集落时,观察到生长量增加了4倍。
2、在单克隆生长情况方面,在第0天、第7天和第14天对96孔培养板进行分析时,确认WOLF G2导致了74±2.9%的单克隆生长,与有限稀释法制备的板中20±7.6%相比,提高了3.75倍(图3)。
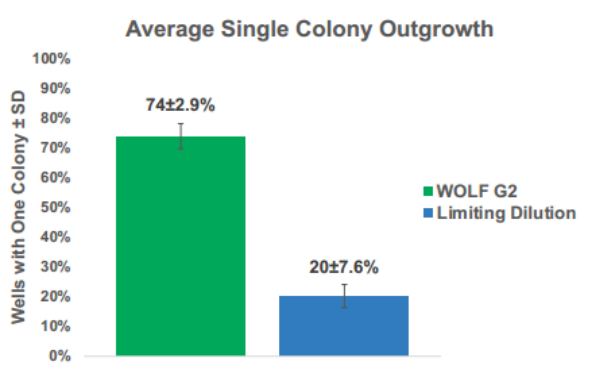
图3. 与有限稀释法相比,使用WOLF G2进行单细胞克隆时的平均单细胞沉积:与极限稀释相比,用于单细胞克隆的WOLF G2产生了3.75倍的孔,其中含有源自单个细胞的单个菌落。
3、单克隆性通过在第0天、第7天和第14天使用Celigo分析细胞,追溯确认检测到的颗粒实际上是细胞,并通过观察它们生长成菌落来验证单克隆性。同时,多天的测量也确保了含有菌落的孔确实来自单个细胞(图4)。
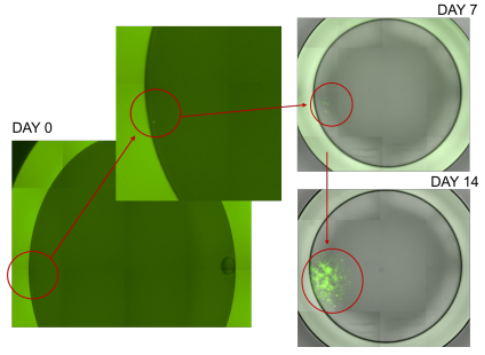
图4. 在第0天、第7天和第14天显示单细胞生长的代表性孔:在使用WOLF G2和N1进行单细胞分选时,第0、7和14天用于确定菌落是否来自沉积在孔中的单个细胞。注意细胞如何保持在相同的位置,并随着细胞的生长而增大。
4、总生长结果与我们之前使用WOLF在没有活性染料的情况下对GFP+ HEK293细胞进行分选的结果一致。先前研究的平均总生长为78±5.5%(图5)[7]。
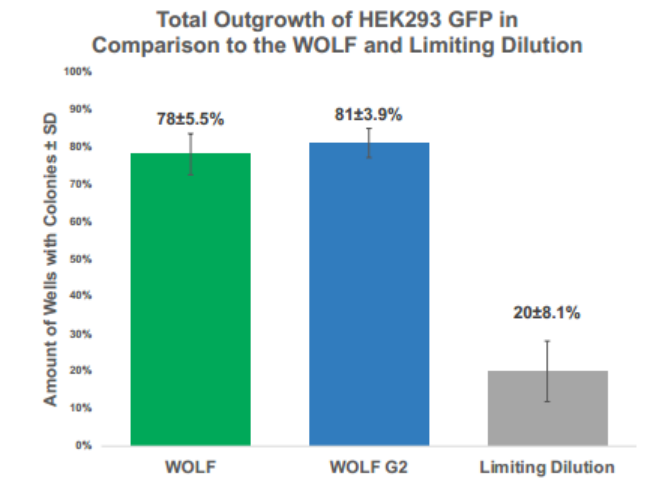
图5. 与WOLF、WOLF G2和有限稀释法相比,HEK293 GFP的平均总产出量在范围内,无论使用哪种WOLF。由于能够产生更多的单细胞克隆,使用WOLF的单细胞分选优于限制稀释。
这项研究表明,WOLF G2与传统的有限稀释法等方法相比,能够确保更高的单克隆性。而且,WOLF G2还有多种激光配置可供选择,如488-405nm和488-561nm等,可用于各种遗传标记、常规染料和新兴化学物质,以满足细胞系的开发以及其他克隆或样品制备工作流程的需求。
同腾睿杰(上海)生物科技有限公司作为Nanocellect中国南区总代理商,为您提供优质的售前售后服务。
联系电话:021-50826962
联系邮箱:sales@ttbiotech.com
1. Zhou, Y., Shaw, D., Lam, C., Tsukuda, J., Yim, M., Tang, D., Louie, S., Laird, M. W., Snedecor, B., & Misaghi, S. (2017). Beating the odds: The Poisson distribution of all input cells during limiting dilution grossly underestimates whether a cell line is clonally-derived or not. Biotechnology Progress, 34(3), 559–569. https://doi.org/10.1002/btpr.2560.
2. Welch, J. T., & Arden, N. S. (2019). Considering “clonality”: A regulatory perspective on the importance of the clonal derivation of mammalian cell banks in biopharmaceutical development. Biologicals, 62, 16–21. https://doi.org/10.1016/j.biologicals.2019.09.006.
3. Quiroz, J., & Tsao, Y. S. (2016). Statistical analysis of data from limiting dilution cloning to assess monoclonality in generating manufacturing cell lines. Biotechnology Progress, 32(4), 1061–1068. https://doi.org/10.1002/btpr.2290
4. Bushnell, T., PhD. (2021, March 11). 3 Reagents For Identifying Live, Dead, And Apoptotic Cells By Flow Cytometry. Cheeky Scientist. https://expert.cheekyscientist.com/3-reagents-for-identifying-live-dead-and-apoptotic-cells-by-flow-cytometry/.
5. Edward, R. (2014, May 1). Compensation-free dead cell exclusion: multi-beam excitation of the far-red DNA binding viability dye DRAQ7. (TECH2P.873). The Journal of Immunology. https://www.jimmunol.org/content/192/1_Supplement/135.4
6. Cossarizza, A., Chang, H., Radbruch, A., Acs, A., Adam, D., Adam-Klages, S., Agace, W. W., Aghaeepour, N., Akdis, M., Allez, M., Almeida, L. N., Alvisi, G., Anderson, G., Andrä, I., Annunziato, F., Anselmo, A., Bacher, P., Baldari, C. T., Bari, S., . . . Zychlinsky, A. (2019). Guidelines for the use of flow cytometry and cell sorting in immunological studies (second edition). European Journal of Immunology, 49(10), 1457–1973. https://doi.org/10.1002/eji.201970107.
7. Munoz, A. (2020). Increase HEK293T Single-Cell Clonal Outgrowth Under Standard Conditions. Retrieved from https://nanocellect.com/scientific-content/.




